Attualità
La Dottoressa Li-Meng Yan presenta le prove dell’origine artificiale del COVID-19. Ora i contestatori saranno contenti


L’intervista della dottoressa Li-Meng Yan ha scaldato gli animi ed alcuni (anche rispondendoci su twitter….) hanno chiesto la prova delle sue affermazioni. Bene, l’accademica cinese non ha atteso molto e, dopo aver aperto un account su Twitter, ha iniziato a postare le basi scientifiche delle sue affermazioni.
Chiaramente non sono dei documenti comprensibili a tutto (magari @Chimicoscettico potrebbe darci un’occhiata…) ma cerchiamo di fornire degli estratti comprensibili.
Prima di tutto ha pubblica un link ad un suo paper scientifico dal titolo piuttosto chiaro:
Come rafforzativo ala ricerca posta un suo profilo accademico da cui risultano 13 paper scientifici e la sua affiliazione all’Università di Hong Kong
Ecco alcuni estratti :
L’evidenza mostra che SARS-CoV-2 dovrebbe essere un prodotto di laboratorio creato utilizzando i coronavirus da pipistrello ZC45 e / o ZXC21 come modello e / o spina dorsale. Basandoci sulle prove, postuliamo ulteriormente un percorso sintetico per SARS-CoV-2, dimostrando che la creazione in laboratorio di questo coronavirus è conveniente e può essere realizzata in circa sei mesi.
Ecco la battuta finale estesa:
Il motivo è che il legame del recettore di SARS-CoV-2 Spike non può nascere dalla natura e dovrebbe essere stato creato attraverso l’ingegneria genetica.
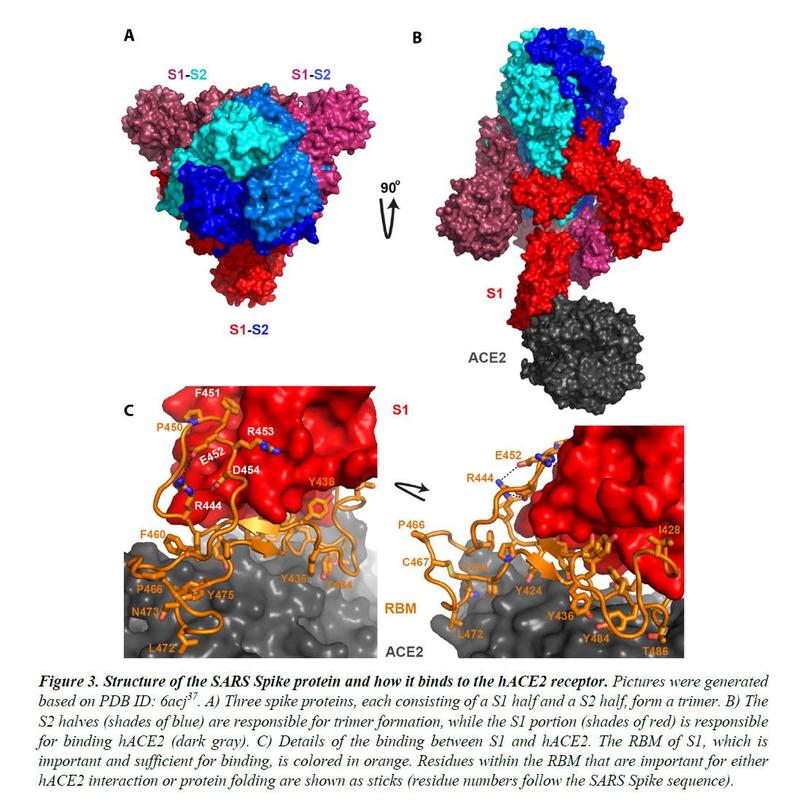
Le proteine Spike decorano l’esterno delle particelle di coronavirus. Svolgono un ruolo importante nell’infezione poiché mediano l’interazione con i recettori delle cellule ospiti e quindi aiutano a determinare la gamma dell’ospite e il tropismo tissutale del virus. La proteina Spike è divisa in due metà (Figura 3). La metà anteriore o N-terminale è chiamata S1, che è completamente responsabile del legame del recettore ospite. In entrambe le infezioni da SARS-CoV e SARS-CoV-2, il recettore della cellula ospite è hACE2. All’interno di S1, un segmento di circa 70 amminoacidi entra in contatto diretto con hACE2 ed è corrispondentemente denominato come la forma legante del recettore (RBM) (Figura 3C). In SARS-CoV e SARS-CoV-2, l’RBM determina completamente l’interazione con hACE2. La metà C-terminale della proteina Spike è denominata S2. La funzione principale di S2 include il mantenimento della formazione di trimeri e, in seguito a scissioni di proteasi successive alla giunzione S1 / S2 e una posizione S2 ‘a valle, mediare la fusione della
Simile a quanto osservato per altre proteine virali, S2 di SARS-CoV-2 condivide un’identità di sequenza elevata (95%) con S2 di ZC45 / ZXC21. In netto contrasto, tra SARS-CoV-2 e ZC45 / ZXC21, la proteina S1, che determina quale ospite (umano o pipistrello) il virus può infettare, è molto meno conservata con l’identità della sequenza di amminoacidi solo del 69%.
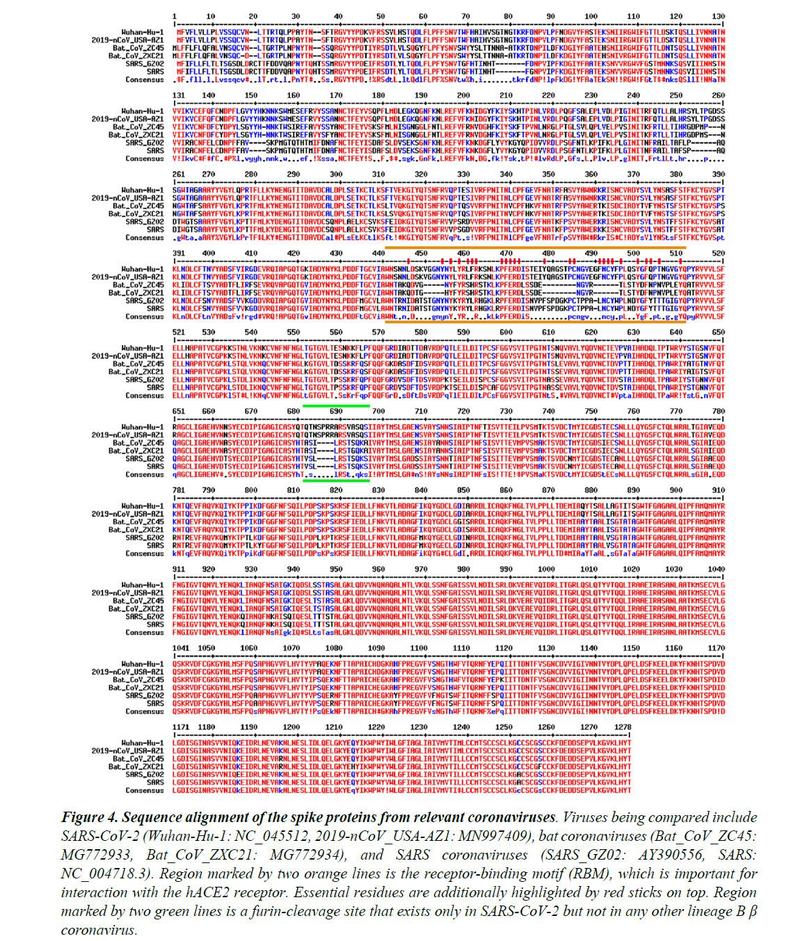
La Figura 4 mostra l’allineamento della sequenza delle proteine Spike da sei coronavirus β. Due sono virus isolati dall’attuale pandemia (Wuhan-Hu-1, 2019-nCoV_USA-AZ1); due sono i sospetti virus template (Bat_CoV_ZC45, Bat_CoV_ZXC21); due sono coronavirus SARS (SARS_GZ02, SARS). L’RBM è evidenziato tra due linee arancioni. Chiaramente, nonostante l’elevata identità di sequenza per i genomi complessivi, l’RBM di SARS-CoV-2 differisce significativamente da quelli di ZC45 e ZXC21. Curiosamente, l’RBM di SARS-CoV-2 assomiglia, in gran parte, all’RBM di SARS Spike. Sebbene questo non sia un “copia e incolla” esatto, un attento esame delle strutture Spike-hACE237,38 rivela che tutti i residui essenziali per il legame di hACE2 o per il ripiegamento delle proteine (bastoncini arancioni nella Figura 3C e ciò che è evidenziato da linee corte rosse nella Figura 4) sono “conservati”.
La maggior parte di questi residui essenziali sono conservati con precisione, compresi quelli coinvolti nella formazione di legami disolfuro (C467, C474) e nelle interazioni elettrostatiche (R444, E452, R453, D454), che sono fondamentali per l’integrità strutturale dell’RBM (Figura 3C e 4) . I pochi cambiamenti all’interno del gruppo dei residui essenziali sono quasi esclusivamente “sostituzioni” idrofobiche (I428àL, L443àF, F460àY, L472àF, Y484àQ), che non dovrebbero influenzare né il ripiegamento delle proteine né l’interazione con hACE2. Allo stesso tempo, la maggior parte dei residui amminoacidici non essenziali sono “mutati” (Figura 4, residui RBM non etichettati con brevi linee rosse). A giudicare dalla sola analisi della sequenza, siamo stati convinti fin dall’inizio che non solo la proteina SARS-CoV-2 Spike si legherebbe a hACE2, ma anche il legame sarebbe stato simile, precisamente, a quello tra la proteina originale SARS Spike e hACE223. I recenti lavori strutturali hanno confermato la nostra previsione.
quindi la scienziata fa due diverse ipotesi che possono essere alla base della nascita “Naturale” del virus. Che questo derivi: 1) un antico evento di ricombinazione seguito da evoluzione convergente o 2) un evento di ricombinazione naturale che si è verificato abbastanza di recente.
Lei quindi passa ad escludere le due ipotesi:
1) “questo processo di evoluzione convergente comporterebbe anche l’accumulo di una grande quantità di mutazioni in altre parti del genoma, rendendo l’identità di sequenza complessiva relativamente bassa. L’identità di sequenza elevata tra SARS-CoV-2 e ZC45 / ZXC21 su varie proteine ( 94-100% di identità) non supportano questo scenario e, quindi, indica chiaramente che SARS-CoV-2 che trasporta un tale RBM non può provenire da un coronavirus di pipistrello simile a ZC45 / ZXC21 attraverso questa rotta evolutiva convergente “.
Quindi elimina l’opzione 2)2) Nel secondo scenario, il coronavirus simile a ZC45 / ZXC21 avrebbe dovuto ricombinare e scambiare di recente il suo RBM con un altro coronavirus che si era adattato con successo per legare un ACE2 animale altamente omologo a hACE2. La probabilità di un tale evento dipende, in parte, dai requisiti generali della ricombinazione naturale: 1) che i due diversi virus condividano una significativa somiglianza di sequenza; 2) che devono co-infettare ed essere presenti nella stessa cellula dello stesso animale; 3) che il virus ricombinante non sarebbe stato eliminato dall’ospite o non avrebbe fatto estinguere l’ospite; 4) che il virus ricombinante alla fine dovrebbe diventare stabile e trasmissibile all’interno della specie ospite.
Per quanto riguarda questo recente scenario di ricombinazione, il serbatoio animale non potrebbe essere pipistrelli perché le proteine ACE2 nei pipistrelli non sono abbastanza omologhe a hACE2 e quindi l’adattamento non sarebbe in grado di produrre una sequenza RBM come visto in SARS-CoV-2. Anche questo serbatoio animale non potrebbe essere umano poiché il coronavirus simile a ZC45 / ZXC21 non sarebbe in grado di infettare gli esseri umani. Inoltre, non ci sono state prove di virus simili a SARS-CoV-2 o SARS-CoV-2 circolanti nella popolazione umana prima della fine del 2019. Curiosamente, secondo un recente studio bioinformatico, SARS-CoV-2 stava bene -adattato per gli esseri umani dall’inizio dell’epidemia.
Scartato l’impossibile, quello che resta, per quanto improbabile, è la verità:
Rimane solo un’altra possibilità di evoluzione naturale, ovvero che il virus simile a ZC45 / ZXC21 e un coronavirus contenente un RBM simile alla SARS potrebbero essersi ricombinati in un ospite intermedio in cui la proteina ACE2 è omologa a hACE2. Diversi laboratori hanno riferito che alcuni dei pangolini della Sonda contrabbandati in Cina dalla Malesia erano portatori di coronavirus, il cui dominio di legame al recettore (RBD) è quasi identico a quello di SARS-CoV-227-29,31. Hanno quindi proseguito suggerendo che i pangolini sono il probabile ospite intermedio per SARS-CoV-227-29,31. Tuttavia, recenti rapporti indipendenti hanno trovato difetti significativi in questi dati . Inoltre, contrariamente a questi rapporti, nessun coronavirus è stato rilevato nei campioni di pangolino Sunda raccolti per oltre un decennio in Malesia e Sabah tra il 2009 e il 2019. Uno studio recente ha anche mostrato che il RBD, che è condiviso tra SARS-CoV -2 e il pangolino coronavirus riportato, si lega a hACE2 dieci volte più forte rispetto al pangolino ACE22, escludendo ulteriormente i pangolini come possibile ospite intermedio. Infine, uno studio in silico, pur riecheggiando l’idea che i pangolini non siano probabilmente un ospite intermedio, ha anche indicato che nessuna delle proteine ACE2 animali esaminate nel loro studio ha mostrato un potenziale di legame più favorevole alla proteina SARS-CoV-2 Spike rispetto a hACE2 . Quest’ultimo studio ha virtualmente esentato tutti gli animali dal loro sospetto ruolo di ospite intermedio, il che è coerente con l’osservazione che SARS-CoV-2 era ben adattato per l’uomo dall’inizio dell’epidemia. Ciò è significativo perché questi risultati suggeriscono collettivamente che nessun ospite intermedio sembra esistere per SARS-CoV-2, il che per lo meno diminuisce la possibilità che un evento ricombinante si verifichi in un ospite intermedio.
Dato che RBM impone completamente il legame con hACE2 e che il legame SARS RBM-hACE2 era completamente caratterizzato da strutture ad alta risoluzione (Figura 3), questo scambio solo RBM non sarebbe più rischioso dello scambio completo Spike. In effetti, la fattibilità di questa strategia di scambio RBM è stata dimostrata. Nel 2008, il gruppo del dottor Zhengli Shi ha scambiato un RBM della SARS nelle proteine Spike di diversi coronavirus di pipistrello simili alla SARS dopo aver introdotto un sito di restrizione in un gene spike ottimizzato per il codone (Figura 5C). Hanno quindi convalidato il legame delle proteine Spike chimeriche risultanti con hACE2. Inoltre, in una recente pubblicazione, l’RBM di SARS-CoV-2 è stato scambiato nel dominio di legame del recettore (RBD) di SARSCoV, risultando in un RBD chimerico completamente funzionale nel legare hACE2 (Figura 5C) . Sorprendentemente, in entrambi i casi, i segmenti RBM manipolati assomigliano quasi esattamente all’RBM definito dalle posizioni dei siti EcoRI e BstEII (Figura 5C). Sebbene i dettagli sulla clonazione manchino in entrambe le pubblicazioni39,47, è concepibile che i siti di restrizione effettivi possano variare a seconda del gene spike che riceve l’inserimento RBM, nonché della convenienza nell’introdurre siti di restrizione unici nelle regioni di interesse. È interessante notare che l’autore corrispondente di questa recente pubblicazione, il Dr. Fang Li, è stato un collaboratore attivo del Dr. Zhengli Shi dal 2010. Il dottor Li è stata la prima persona al mondo ad aver chiarito strutturalmente il legame tra SARS-CoV RBD e hACE238 ed è stato il principale esperto nella comprensione strutturale delle interazioni Spike-ACE2. La sorprendente scoperta di siti di restrizione EcoRI e BstEII alle due estremità del SARS-CoV-2 RBM, rispettivamente, e il fatto che la stessa regione RBM sia stata scambiata sia dal Dr. Shi che dal suo collaboratore a lungo termine, rispettivamente, utilizzando i metodi di digestione con enzimi di restrizione sono improbabili come una coincidenza. Piuttosto, è la pistola fumante che dimostra che l’RBM / Spike di SARS-CoV-2 è un prodotto della manipolazione genetica “.
Pare che i cinesi abbiano anche provato a nascondere le prove…
Sebbene possa essere conveniente copiare la sequenza esatta dell’RBM della SARS, sarebbe un segno troppo chiaro di progettazione e manipolazione artificiali. L’approccio più ingannevole sarebbe quello di modificare alcuni residui non essenziali, preservando quelli critici per il legame. Questo progetto potrebbe essere ben guidato dalle strutture ad alta risoluzione (Figura 3) 37,38. In questo modo, quando la sequenza complessiva dell’RBM sembrerebbe essere più distinta da quella dell’RBM SARS, la capacità di legare hACE2 sarebbe ben preservata. Riteniamo che tutti i residui cruciali (residui etichettati con bastoncini rossi nella Figura 4, che sono gli stessi residui mostrati nei bastoncini nella Figura 3C) avrebbero dovuto essere “conservati”. Come descritto in precedenza, mentre alcuni dovrebbero essere la conservazione diretta, alcuni dovrebbero essere passati a residui con proprietà simili, che non interrompono il legame con hACE2 e potrebbero persino rafforzare ulteriormente l’associazione [ZH: cioè, il virus è stato armato e potenziato]. È importante sottolineare che le modifiche potrebbero essere state apportate intenzionalmente a siti non essenziali, rendendolo meno simile a un “copia e incolla” del SARS RBM.
Quindi non sono c’è stata una manipolazione genetica, ma gli autori han fatto tutto il possibile per cercare di nasconderla. Una situazione veramente imbarazzante per il governo cinese se venisse scientificamente confermata a livello internazionale e che giustificherebbe tutte le accuse fatte.